-
SSR即简单序列重复(Simple sequence repeats)又称为微卫星(Microsatellites)[1-2],是以如(CA)n,(TG)n,(GGC)n等少数几个核苷酸为单位的串联重复DNA序列,其中n代表重复次数,重复次数从几个到几十个不等。不同等位基因之间的重复数存在着显著差异,并且重复序列两端多是相对保守的单拷贝序列,可通过设计特异性引物对基因组总DNA进行PCR扩增,扩增片段的长度多态性可用作分子标记[3]。SSR分子标记是群体遗传结构分析、品种遗传多样性研究、品种保护与纯度鉴定、交配系统分析的有效工具,也被认为是信息含量高、多态性高;稳定性好,重复性好;分辨率高、揭示力强的分子标记。
竹类植物是禾本科中最大的自然类群,全世界约有88属1 400种,中国有34属近550种,集材用、笋用和观赏等众多用途,是重要的园林树种之一[4-5]。竹类植物作为一类生物学特性特殊的植物,近年来,虽然SSR分子标记技术在其种质鉴定、种属分类及系统发育等方面有所应用,但是竹类植物中开发的SSR引物较少,检测的SSR位点不足以满足种质鉴定及群体遗传研究的需要,因而需要开发更多的竹类植物SSR引物,来扩大竹类植物种质鉴定的数量和范围及进行系统分类、群体遗传等研究。本文综述了竹类植物中的SSR引物开发方法及研究进展,为进一步研究竹类植物种质资源的群体遗传学提供技术支持。
-
由于SSR标记是根据微卫星两侧的保守序列设计引物的,因此,不同物种之间的有些SSR引物是可以通用的。在早期的研究中,因没有开发出竹类植物SSR引物,竹类植物SSR分子标记也处于摸索阶段,学者们便利用竹类植物与禾本科其他植物的亲缘关系,将水稻、小麦、高粱等禾本科植物的分子数据运用到竹类研究中,是研究竹类植物SSR标记的有效途径之一。在国内的研究中,具有代表性的是将水稻SSR引物数据用于竹子中。李淑娴等[6]通过水稻SSR引物从分子方面分析青篱竹属及其相关属的亲缘关系,陈淑云等[7]利用水稻和甘蔗SSR引物转移至竹子中,开发出42对SSR引物(水稻34对、甘蔗8对)用于竹子的表型和遗传多样性分析。国外也有相似的研究,用禾本科植物的SSR引物检测竹子的遗传变异、竹种鉴定、遗传多样性[8-9]。国内外文献表明,禾本科植物的SSR引物适用于竹类植物研究,说明SSR引物在同科不同属或者更近的物种间具有一定的通用性。因此,使用近缘种的引物可以很好地用于竹类植物的系统发育和遗传多样性研究。
-
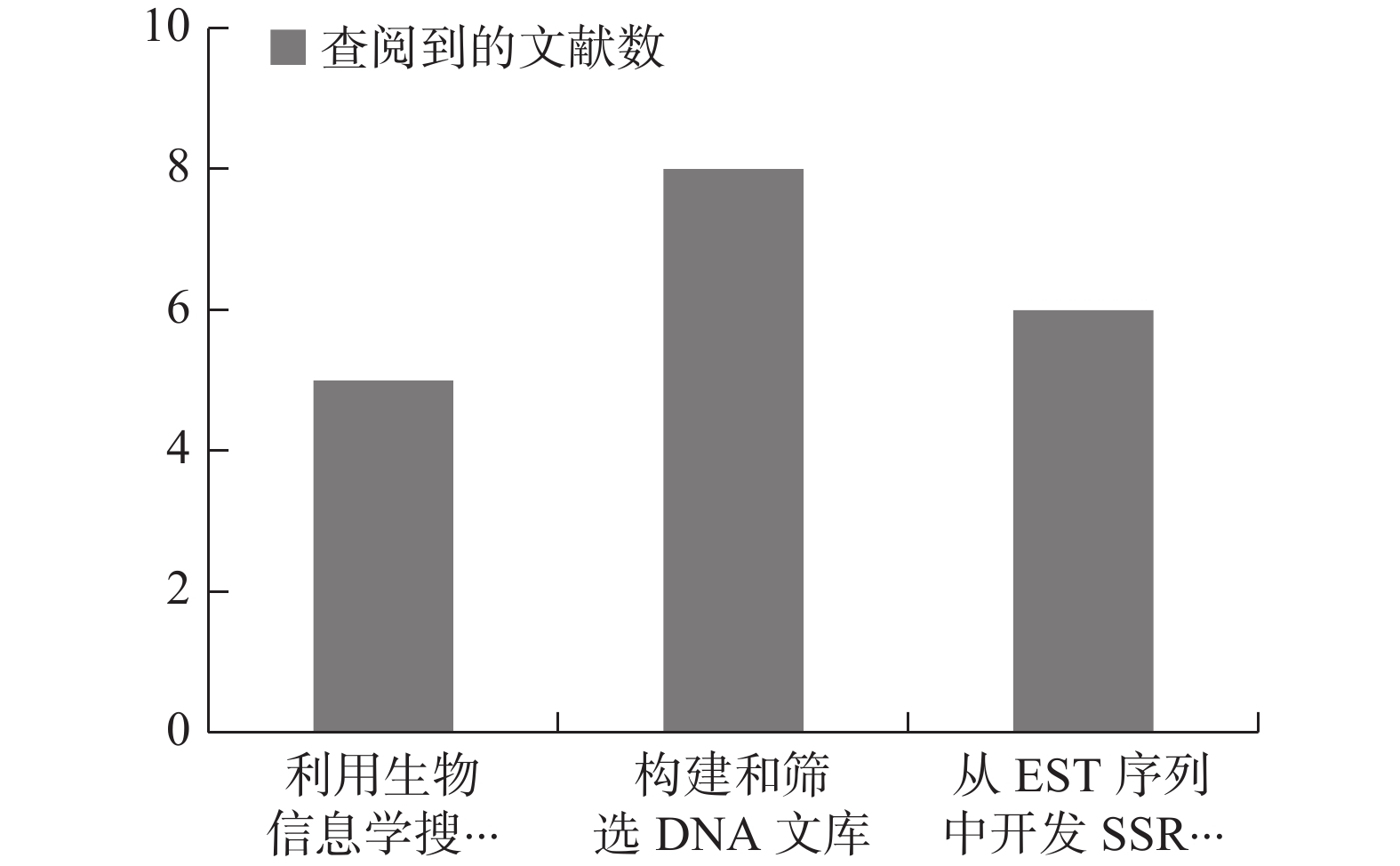
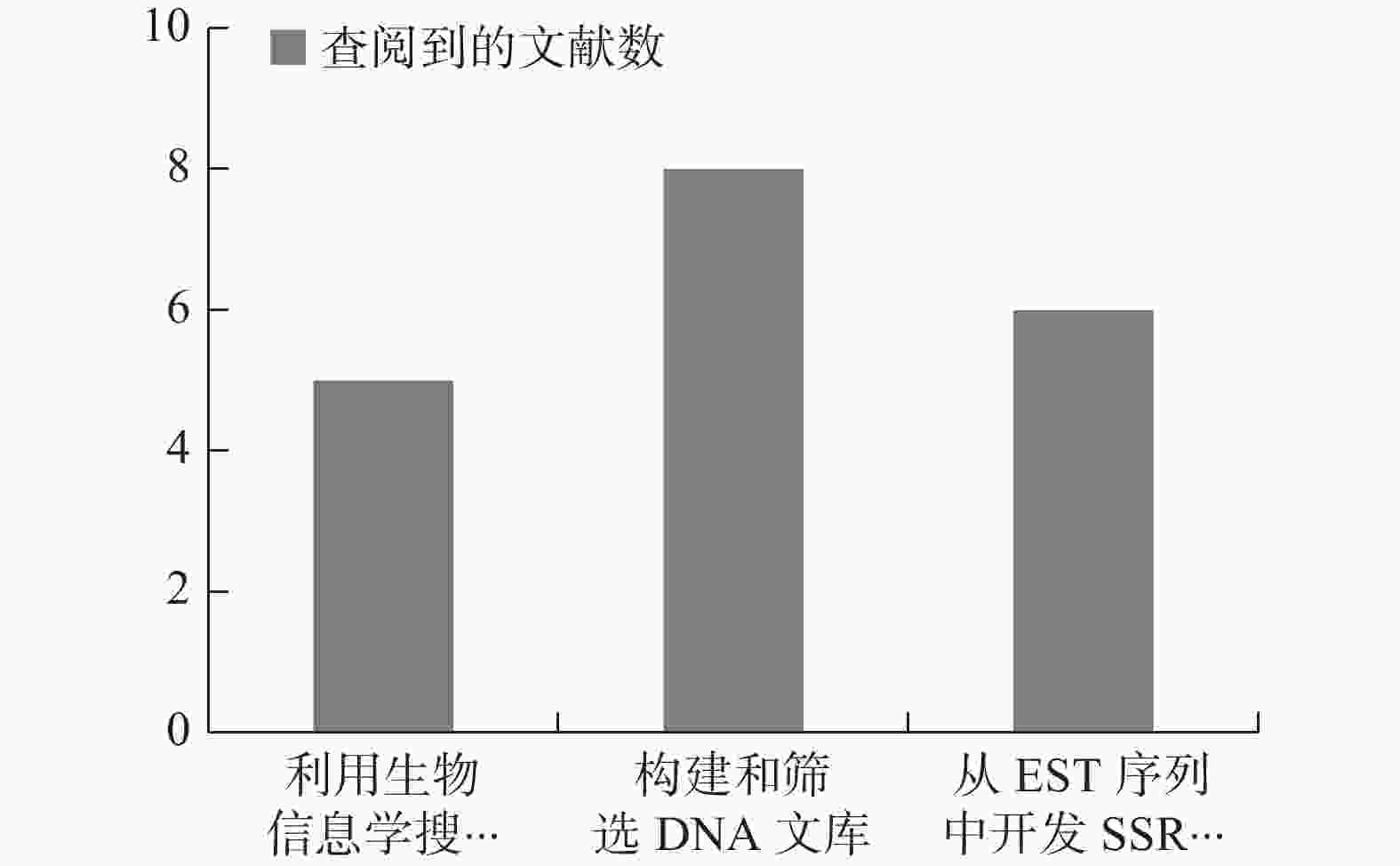
开发SSR引物的前提是要知道SSR位点两侧的DNA序列。目前,对于竹类植物SSR引物开发策略,从搜索的文献资料来看,如图1所示,常用主要有3种:利用生物信息学搜索数据库、构建和筛选基因组文库、从EST序列中开发SSR引物。
-
在DNA数据库中已经存在大量的基因组序列,可以先通过SSR搜索软件获得SSR位点信息,这些搜索软件包括在线的SSRIT(http://www.gramene.org/ab/searches/ssrtool)Sputnik(http://cbi.labri.fr/outils/Pise/sputnik.html)及可本地下载的软件比如DNAman、misa、ssrhunter等[10]。然后根据SSR位点两侧的核酸序列信息,利用软件设计相应的引物,常用的引物设计软件有primer premier 5.0 和oligo 6.0,也可以利用在线设计软件如primer 3.0 进行引物设计[11]。
已开发出相关基因组数据库有很多种,如国际三大核苷酸数据库Gene Bank/NCBI、EMBL以及DDBJ等。也有专门的微卫星DNA引物数据库如Molecular Ecology Notes。卢江杰等[12, 13]通过搜索Gen Bank毛竹序列,开发了19对SSR引物,并用这些引物研究了毛竹11个变型的遗传多样性。通过搜索Gen Bank中的毛竹序列,毛竹已成为SSR引物开发最多的竹种。
-
构建基因组文库发筛选SSR引物是最传统,也是最经典的方法。它首先需要构建所研究对象的一个小片段插入的基因组文库,然后用含有微卫星序列的探针与之杂交筛选文库,然后挑选阳性克隆进行测序,再根据SSR两端的侧翼序列设计引物,最后用该引物对相应的SSR座位作定性分析[14]。这种方法需要进行多次筛选,并且需要测序,具有工作量大、费时费力的缺点。
在竹类植物中,最早的研究是Nayak和Rout等[15]对簕竹属的印度刺竹(Bambusa arundinacea)采用传统的基因组文库构建和Southern杂交的传统方法,获得了6对SSR引物,其中仅3对具有多态性,分析了18个亲缘竹种间的遗传多样性。Kaneko等 [16]利用构建文库法,针对澳大利亚北部特有竹种Bambusa arnhemica开发了9对SSR引物,分析了该种4个群体95个体的开花现象。
-
为了提高筛选工作效率,人们开发了利用富集步骤筛选微卫星标记的方法—建立和筛选微卫星富集文库法。其方法主要有两种:一种是利用含微卫星序列的PCR引物进行PCR富集。另一种是用含微卫星序列的探针进行杂交富集。实质上,也就是在筛选小插入片段基因组文库的基础上再增加一次筛选。或者先用PCR富集,再用探针杂交筛选;或者两次都用杂交筛选,总之,经过这样两次筛选,获得SSR克隆的效率大大提高。富集文库的建立,有效避免了克隆的筛选,缩短了筛选时间,适宜于大规模的开发,可以在不同程度上降低开发成本。在竹类植物中,利用磁珠富集法来开发SSR引物应用较为广泛。
从图1可知,从已发表的文献资料来看,采用构建基因组文库开发SSR引物的文献较多。其中,利用磁珠富集法所开发的毛竹SSR引物的文章就有5篇[12, 17-20]。另外,董玉然等[21]在磁珠富集法的基础之上,采用基于AFLP技术的磁珠富集快速分离技术(FIASCO)(即按照AFLP的方法进行酶切、连接、扩增后再利用磁珠杂交富集含有微卫星的DNA片段)开发了16对巨龙竹SSR引物,并用这些引物分析了巨龙竹的遗传多样性。
-
传统的基因组SSR标记开发过程较慢,需要进行基因组文库的构建、重复序列克隆的识别和筛选、测序、引物设计等环节,不仅费时耗力,成本也很高。近年来通过cDNA文库测序得到的表达序列标签EST(Expressed sequence tag,EST)发展十分迅速,在GenBank中平均每天都有上万个EST公布,因而从EST序列中可迅速寻找到EST-SSR标记[22]。由于EST代表具有一定功能基因或基因的一部分,使得EST-SSR具有较强的种属通用性。
李桃等[23]搜索了NCBI、EMBL中的竹类EST序列,通过比对和SSR搜索后设计了35对引物,并将16个属的39种竹类植物分为丛生竹和散生竹两大类群。Sharma等[24]基于NCBI中Bambusa oldhamii的EST序列开发了13 对EST-SSR引物,其中能扩增目的片段的10 对引物在 25 个竹种中的通用性进行了研究,通用率为30% ~ 100%不等,多态信息含量(PIC)约0.54,由于SSR位点代表转录区域及种间高通用性,均表明EST-SSR可用于竹种功能及遗传分析。目前,在竹类植物研究中,从EST序列中获得的SSR标记在系统分类、竹种间的通用性[25]、竹种鉴定[26-27]、遗传多样性[28]等领域有广泛应用。
-
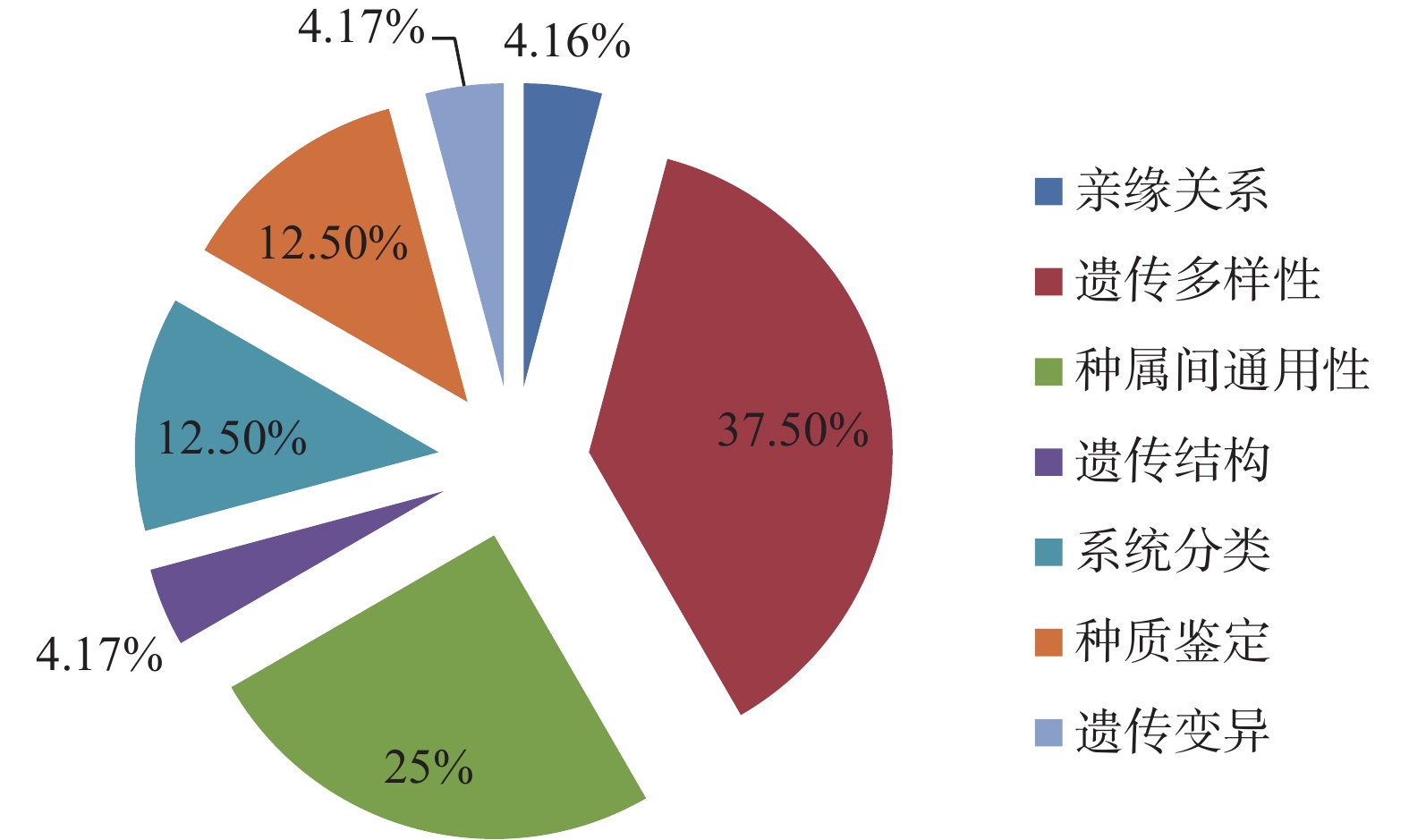
随着测序技术的发展,SSR分子标记因其操作简单和共显性,在竹类植物中已经有了广泛的应用。如图2所示,从现有已发表的国内外文献来看,SSR分子标记在竹类植物中的应用主要集中遗传多样性分析和种属间的通用性研究,分别占所搜集文献的37.5%和25%;竹子分类系统和种质鉴定次之,均占12.5%;亲缘关系分析、遗传变异、遗传结构等领域的应用较少,分别占4.16%、4.17%、4.17%。
在植物遗传育种研究中,叶绿体SSR标记、细胞核SSR标记已在遗传结构分析[29]、遗传多样性和亲缘关系[30]等研究中有了广泛应用,对于竹类植物来说,开发这些SSR标记任重而道远,仍然需要致力于竹类植物研究的学者们去不断探索。
-
随着分子生物学实验技术的发展和改进,SSR分子标记已得到了广泛应用,新的SSR标记开发策略也不断地被提出。上述所提到的几种SSR引物开发策略,各有各的优势,因此,在进行SSR引物时,要根据实验条件和材料要求,选择合适的开发策略是成功开发SSR引物的关键所在。
目前,SSR标记在植物中的应用主要包括通用性研究、遗传多样性与种质鉴定研究、构建遗传连锁图谱、基因定位与克隆等,通用性、遗传多样性与种质鉴定研究在竹类植物中已经有所应用,且从目前的文献资料来看,以遗传多样性研究为主。在竹类植物中构建遗传图谱、基因定位与克隆发展相对较慢,然而,国内外学者们,利用SSR构建遗传连锁图谱在水稻[31-32]、小麦[33]、棉花[34]等农作物中已经取得了突破性的进展,主要作物的遗传图谱的成功构建,也将为基因的精细定位与图位克隆奠定坚实的基础。例如:控制玉米种子含油量和胚含油量的基因DGAGl-2在初步定位时,就是被定位在两个SSR标记之间,最终通过图位克隆获得了功能基因[35]。根据毛竹基因组序列草图,60%的毛竹基因组为重复序列所覆盖[36],说明SSR标记在竹类植物研究中有着巨大的潜力。因此,相信随着分子标记技术的发展与不断创新,构建竹类植物的遗传图谱及基因定位与克隆也只是时间的问题。
竹类植物是比较特殊的一类,其染色体数目较多且不稳定,普遍存在多倍性、非整倍性、混倍性和染色体结构变异的现象。竹类植物染色体结构和基因组特性方面的研究尚未见报道,如果能够了解竹类植物染色体上SSR序列的分布情况,从而探索微卫星在竹类植物染色体上的变化,将有利于进一步推断竹类植物的染色体结构和进化过程,为竹类植物的遗传学研究奠定基础。
Exploitation Strategies and Applications of SSR Primer in Bamboo Plants
doi: 10.12172/201909240001
- Received Date: 2019-09-24
- Available Online: 2019-12-20
- Publish Date: 2020-02-27
-
Key words:
- Bamboo plants /
- SSR Primer /
- Exploitation strategy /
- Application
Abstract: Microsatellites (SSR) refer to DNA sequences that are repeated in series with nucleotides as units, and widely distributed throughout the genome of eukaryotes. SSR marker is a molecular maker technology based on DNA length polymorphism, which is widely used in the fields of population genetic structure analysis, establishment of genetic map, gene location, genetic relationship, and so on. In this paper, the exploitation methods and applications of SSR primers in bamboo plants were reviewed, aiming to provide useful information for SSR molecular marker in bamboo plants.



















 DownLoad:
DownLoad: