-
遗传多样性是探究不同群体间、群体内不同个体间的遗传变异程度的基础[1],能指导物种多样性、生态系统多样性的相关研究[2]。一般情况下,具有更高的遗传多样性的物种,对环境的适应能力更强。群体的遗传多样性越高,种群内不同个体间的生理生化、表型特征就越丰富,能够适应的环境情况就越复杂[3]。因此,研究物种的遗传多样性水平可以分析其当下的生存状况与发展潜力,筛选出环境适应力强的种质资源,为发掘优良种质资源、保护树种资源奠定基础[4-5]。如张海东[6]综合考虑濒危保护树种望天树不同家系幼苗的表型差异和遗传多样性特征,筛选出了优良家系。蒋迎红[7]研究珍稀濒危野生树种海南风吹楠的生物学特性、群落特征和种群遗传多样性,揭示了其濒危机制与幼苗更新、种子萌发特征的关联性,并提出相应保护培育策略。李娟[8]对5个闽楠野生种群共96个样本的遗传多样性和种群遗传结构进行分析,其结果指导了闽楠种质资源保护、利用工作。分子标记技术是利用基因片段差异建立的检测技术,是遗传变异的直接反映[9]。SSR(Simple Sequence Repeat,简单重复序列),又称之为微卫星,在生物遗传多样性、资源亲缘关系、品种鉴定、遗传图谱构建等方面广泛应用[10]。例如,赵盼[11]运用SSR标记,分析出千年桐种质资源的遗传多样性水平和遗传结构。熊远芳[12]筛选出适用闽楠的23对SSR多态性引物,研究了其遗传多样性研究,为后续的分子标记辅助育种奠定了基础。刘丹[13]开发闽楠基因组SSR和EST-SSR,应用于闽楠优良野生群体的遗传多样性研究,该分子标记具有稳定、多态性高等优点,在楠木近缘属具有通用性。
楠木(Phoebe zhennan),俗称桢楠,为樟科楠属大乔木,是国家二级保护树种,其木材结构细腻、纹理美观,抗虫蛀蚀,不易腐蚀,为建筑、家具、雕刻等领域的高档材料,位居四大名木之首[14]。楠木木材颜色多呈浅黄色,具有黄褐色色泽,达到一定龄级的大径级材还可能具有金黄色绢丝状光泽,是当前行业公认的能够形成“金丝楠木”的最优树种之一,在珍贵木材产业中具有广阔的应用和市场前景[15]。早期楠木的研究主要集中于栽培技术等方面,近年来,楠木遗传多样性研究与种质资源的评价才逐渐受到重视。张炜[14]率先使用AFLP分子标记技术对6个省92株楠木母树进行了多样性分析,将其划分为3个遗传群体;李娟等[16]使用ISSR分子标记技术对楠木、闽楠、细叶楠、紫楠等7个树种/群体35份样品进行了遗传聚类分析,发现楠木与闽楠的遗传相似系数较高。在此基础上,张群[17]与Zhu[18]分别使用SSR分析标记与核SSR、叶绿体SSR、表型标记结合的方法对5个省份102份楠木优树家系材料进行了遗传多样性分析,分别构建了60份、64份核心种质的分子身份信息。虽然楠木的遗传多样性研究取得了一系列成果,但目前的研究仅涉及四川省成都、雅安部分楠木种源(家系),作为楠木主要产区,四川省的楠木资源较为丰富,许多地区种源(家系)的遗传背景尚不清楚。鉴于此,课题组利用前期开发的14对SSR分子标记对四川省10个主要楠木种源39株母树的半同胞家系进行遗传多样性和遗传结构分析,解析其遗传关系,力求为四川省楠木优异基因资源的发掘和杂交亲本的选配提供理论参考。
-
选择具有代表性的四川省内的10个种源地楠木优树的39个半同胞家系(表1)作为供试材料。于2020年对四川省内的10个种源地39株楠木优树进行采种,次年3月在玉蟾山国家林草长期科研基地进行播种育苗。2022年采集对各家系嫩叶进行混合采样,加入硅胶脱水带回实验,于−80 ℃冰箱中保存备用。
种源编号
Provenance number家系编号
Families种源地
Provenance经度/°
Longitude/°纬度/°
Latitude/°CDct F1-F2 成都青羊杜甫草堂 104.02971 30.65978 CDcz F3-F4 成都崇州 103.67285 30.63014 CDdy F5-F9 成都大邑 103.52075 30.58738 CDql F10-F19 成都邛崃 103.46283 30.41489 LSems F20-F23 乐山峨眉山 103.48440 29.60117 LZfws F24-F31 泸州合江法王寺 105.83520 28.81005 LZnx F32-F33 泸州纳溪 105.37255 28.77343 MShy F34-F35 眉山洪雅 103.37313 29.90661 YAyc F36-F37 雅安雨城 103.03305 30.00531 YAyj F38-F39 雅安荥经 102.84652 29.79402 Table 1. The number and collection site of 39 families
-
采用改良 CTAB 法快速提取楠木基因组DNA。
(1)取保存的楠木叶片,置于液氮中并加入适量PVP,通过研磨成粉末状;取0.1~0.4 g(根据样品数量定)研成粉末的样品,加入55 ℃预热的2×CTAB提取缓冲液1~4 mL(与样品粉末比例为1∶10),加入40~160 uL 4%体积浓度β-巯基乙醇,再加入10~40 uL RNase A,充分振荡混匀,55 ℃水浴20 min;
(2)加入等体积氯仿,抽提多酚和多糖物质,进行轻力度翻转混匀,混匀至无颜色变化为止,1000×g离心10 min,过滤得到上清液;
(3)测量上清液体积,以24∶1的比例混合氯仿和异戊醇,加入等体积混合溶液,同上述方法进行翻转混匀,抽提叶绿体以及其他细胞残渣,再以10000×g离心10 min;
(4)提取上清液,加入等体积−20 ℃预冷的异丙醇,充分混匀,析出DNA后以10000×g离心2 min,倒去全部液体,使用4 mL 70%酒精将DNA沉淀冲下;
(5)使用1.5 mL 70%酒精重复清洗DNA沉淀两次,再使用1 mL100%酒精清洗,清洗后放置通风橱中吹干;
(6)加入200 uL无菌水溶解DNA(无菌水先预热以提高DNA溶解);
(7)使用0.8%琼脂糖凝胶电泳检测提取的DNA质量。
-
(1)以前期研究筛选的14对(表2)条带清晰、稳定且重复性好、多态性高的引物进行PCR扩增。PCR扩增体系参考杨汉波等[19]的桢楠SSR分子标记应用专利。
位点Locus 引物序列Primer sequence (5’-3’) 预期产物大小
Predicted size/bpF(正向引物) R(反向引物) MN-g30 AGAGATGTACCTCGCTGCGT GAGAGCCCATCATGACCAAT 156 MN-e96 TGGGTTTAGCCGTCACTACC TCACTGCCTTGGCTCTGTAA 107 MN-g3 TTGAGGGGGAATGTTGAGAG TGAAGGCAACTGATCACAAGA 248 MN-g5 AAATGGTTGGGTCAAGTTCG GTGCCCATATACCCGATGAC 325 MN-g18 GAAGGTCCTCCTATCCTGCC AATCCGGCTGATACTTCTGC 153 Unigene29601 GCAGGTATCTGTTGTGTCTTC CATTCGTCTTCTTGGAGTCATC 287 SSR17 AACAGGAGAAGGGAAGCAATGG GCCTTCAGCAATGGTGTCGG 375 SSR3 GCTAGAGCTCAAAGGATCCC GGTGGTGATTGGACTGGTAGGAG 352 SSR1 TCGATTTGCAGAAGATAAGCC GGGGTAGAAAAGTGAAAGAGTTG 344 SSR19 CGCATCTCGCCATCCTAC GGTTTACGGTGACCTTCG 449 SSR20 AACGGAGGATGAGATTTATAGG GGAAGAAGAATTAGAAGACGC 436 SSR21 AGTAATACCAGCAGTACCAGTC CAGATAGCATCAGAAGCAGA 437 SSR16 ATGTGGGATAGCCTTGATGTACT CAAATGCCAACCCAATGT 126 PZmk03 GGTTTTCAAGACCGGGGCTA CATGGAGTCCGGGGAAATCC 444 Table 2. The information of SSR used in the genetic analysis of Phoebe zhennan
(2)PCR反应体系共25 uL:上下游引物各1 uL,DNA模板1 uL,PCR mix 12.5 uL,9.5 uL的ddH2O。
(3)PCR反应程序为:94 ℃预变性4 min;共35个扩增循环(扩增循环为94 ℃变性30 s,56~60 ℃复性30 s,72 ℃延伸1 min);扩增循环后72 ℃延伸5 min。
(4)PCR扩增产物使用Fragment Analyzer全自动毛细管电泳分析仪进行检测。
-
使用Excel统计毛细管电泳分析结果,通过Cervus 3.0.7软件计算各位点多态信息含量(PIC),利用GenAlEx 6.502软件对不同种源样品进行遗传多样性分析,计算遗传参数。
-
采用STRUCTURE软件对毛细管电泳分析结果进行种源(家系)遗传结构分析。分析参数iterations和burn-in period均设为10000,确定K值取值范围为1—10,每个K值进行个体分组分析。运行100次后,在Structure Harvester网站上确定最佳K值。利用GenAlEx 6.502软件计算家系间遗传距离,并进行主坐标分析(PCoA),同时利用Powermarker和MEGA软件绘制基于遗传距离的UPGMA树状聚类图。
-
14对SSR引物在39个楠木家系中检测出178个等位基因(Na),在3(MN-e96)~29(SSR16)之间变化,平均为12.714(表3)。有效等位基因数(Ne)在1.507(MN-e96)~16.781(SSR16)范围之间,平均值为7.058。Shannon’s 信息指数(I)在0.620(MN-e96)~3.083(SSR16)之间变化,平均值为1.933。期望杂合度(He)与观测杂合度(Ho)分别在0.336(MN-e96)~0.940(SSR16)之间和0.000(MN-g18、MN-g30)~0.943(SSR16)之间,平均分别为0.764和0.558。SSR1、SSR17、SSR3、SSR19、SSR16、MN-e96、SSR21、位点的Ho均大于He,说明这7个位点都具有比较高的杂合度。MN-g30位点(0.939)、SSR16位点(0.937)和MN-g3位点(0.919)的PIC 数值均接近1,多态性最高,所有位点的PIC平均值为0.746。
位点Locus 等位基因数Na 有效等位基因数Ne Shannon’s信息指数I 观测杂合度Ho 期望杂合度He 固定指数F 多态信息含量PIC SSR1 12.000 7.243 2.173 0.865 0.862 -0.003 0.848 SSR17 5.000 1.798 0.865 0.447 0.444 -0.008 0.410 SSR3 4.000 3.040 1.218 0.816 0.671 -0.216 0.610 SSR19 5.000 3.281 1.312 0.750 0.695 -0.079 0.642 SSR16 29.000 16.781 3.083 0.943 0.940 -0.003 0.937 SSR21 14.000 7.420 2.231 0.872 0.865 -0.008 0.851 SSR20 18.000 9.570 2.505 0.531 0.896 0.407 0.887 PZmK03 9.000 3.010 1.394 0.324 0.668 0.516 0.614 Unigene29601 7.000 3.956 1.567 0.641 0.747 0.142 0.710 MN-e96 3.000 1.507 0.620 0.343 0.336 -0.019 0.306 MN-g3 26.000 13.038 2.891 0.865 0.923 0.063 0.919 MN-g5 17.000 9.063 2.440 0.417 0.890 0.532 0.880 MN-g18 11.000 6.259 2.045 0.000 0.840 1.000 0.887 MN-g30 18.000 12.844 2.713 0.000 0.922 1.000 0.939 Table 3. The genetic diversity parameters of 14 SSR primers
-
各楠木种源的Na和Ne变化范围分别在4.429(乐山峨眉山种源Lsems)~6.214(成都邛崃种源CDql)和3.758(泸州法王寺LZfws)~4.430(CDql)之间(表4)。观测杂合度(Ho)变化范围为0.920(LZfws)~1.000(成都大邑CDdy、CDql、Lsems)。Shannon’s 信息指数(I)变化范围为1.306(Lsems)~1.511(CDql),平均值为1.368。期望杂合度(He)变化范围为0.646~0.703,LZfws种源最小(0.646),CDql种源最大(0.703)。各种源的Shannon’s信息指数(I)在1.306~1.511之间,表现出较高的遗传多样性水平。
种源Provenances 等位基因数Na 有效等位基因数Ne Shonnon’s信息指数I 观测杂合度Ho 期望杂合度He 固定指数F 成都大邑 4.643 3.897 1.349 0.483 0.684 0.266 成都邛崃 6.214 4.430 1.511 0.559 0.703 0.200 乐山峨眉山 4.429 3.854 1.306 0.595 0.672 0.123 泸州法王寺 4.929 3.758 1.307 0.563 0.646 0.150 注:成都杜甫草堂、成都崇州、泸州纳溪、眉山洪雅、雅安雨城、雅安荥经此6个种源地的家系数不足3个,故不进行种源遗传多样性分析。 Table 4. The genetic diversity of different provenances
-
方差分量比结果显示,供试楠木遗传变异的99%来自种源内,仅1%来源于种源间,说明供试楠木家系的遗传变异主要发生在种源内家系间(表5)。楠木种源间的基因流(Nm)在0.716~7.353之间。近交系数(Fis)在−1.000~−0.286之间,平均为0.054。群体分化指数(Fit)的变化范围是SSR3(−0.241)~MN-g30、MN-g18(1.000),平均值为0.140,说明楠木种源杂合子不足(表6)。
变异来源Source 自由度d.f 离差平方和Sum of squares 方差分量Variance component 方差分量比(%)Percentages variation(%) 种源间Among provenances 3 1.552 0.007 1% 种源内Within provenances 27 13.500 0.500 99% 总和Total 53 24.852 0.507 100% Table 5. The AMOVA among provenances
位点Locus 群体内近交系数Fis 群体间近交系数Fit 群体间分化系数Fst 基因流Nm SSR1 0.054 0.140 0.091 2.510 SSR17 0.038 0.085 0.049 4.867 SSR3 −0.286 −0.241 0.035 6.958 SSR19 −0.210 −0.121 0.073 3.176 SSR16 −0.165 −0.085 0.069 3.384 SSR21 −0.091 −0.040 0.047 5.108 SSR20 0.392 0.459 0.110 2.031 PZmK03 0.455 0.497 0.078 2.975 Unigene29601 0.087 0.117 0.033 7.353 MN−e96 −0.241 −0.148 0.075 3.104 MN−g3 −0.074 −0.009 0.060 3.928 MN−g5 0.517 0.581 0.133 1.636 MN−g18 1.000 1.000 0.143 1.496 MN−g30 1.000 1.000 0.259 0.716 Mean 0.054 0.140 0.091 2.510 Table 6. The genetic differentiation of P. zhennan provenances
-
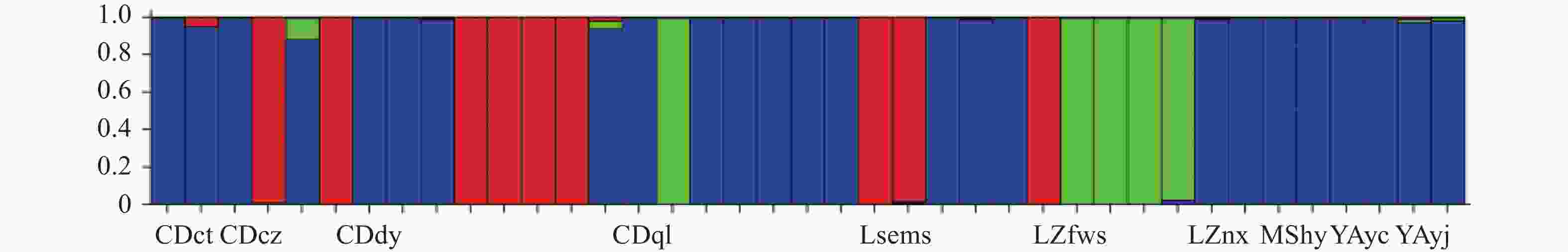
利用STRUCTURE软件对楠木家系进行群体结构分析。当K=3时,ΔK有最大峰值(图1)。即39个家系的遗传成分应该来自相同3个类群,其中成都杜甫草堂、崇州、大邑、邛崃(F1、F2、F3、F5、F7、F8、F9、F14、F15、F17、F18、F19)、乐山峨眉山(F20、F21)、泸州法王寺、纳溪(F24、F25、F26、F32、F33)、眉山洪雅(F34)和雅安雨城、荥经(F35、F36、F37、F38、F39)的25个家系聚为第一类群,成都崇州、大邑、邛崃(F4、F6、F10、F11、F12、F13)、乐山峨眉山(F22、F23)和泸州法王寺(F27)的9个家系聚为第二类群,其余5个家系为第三类群。三个类群都混合了来自成都、泸州的楠木家系,雅安雨城和荥经仅聚类在第一类群中,眉山洪雅只聚类于第三类群中聚类。结果显示类群划分与种源地无明显联系(图2)。
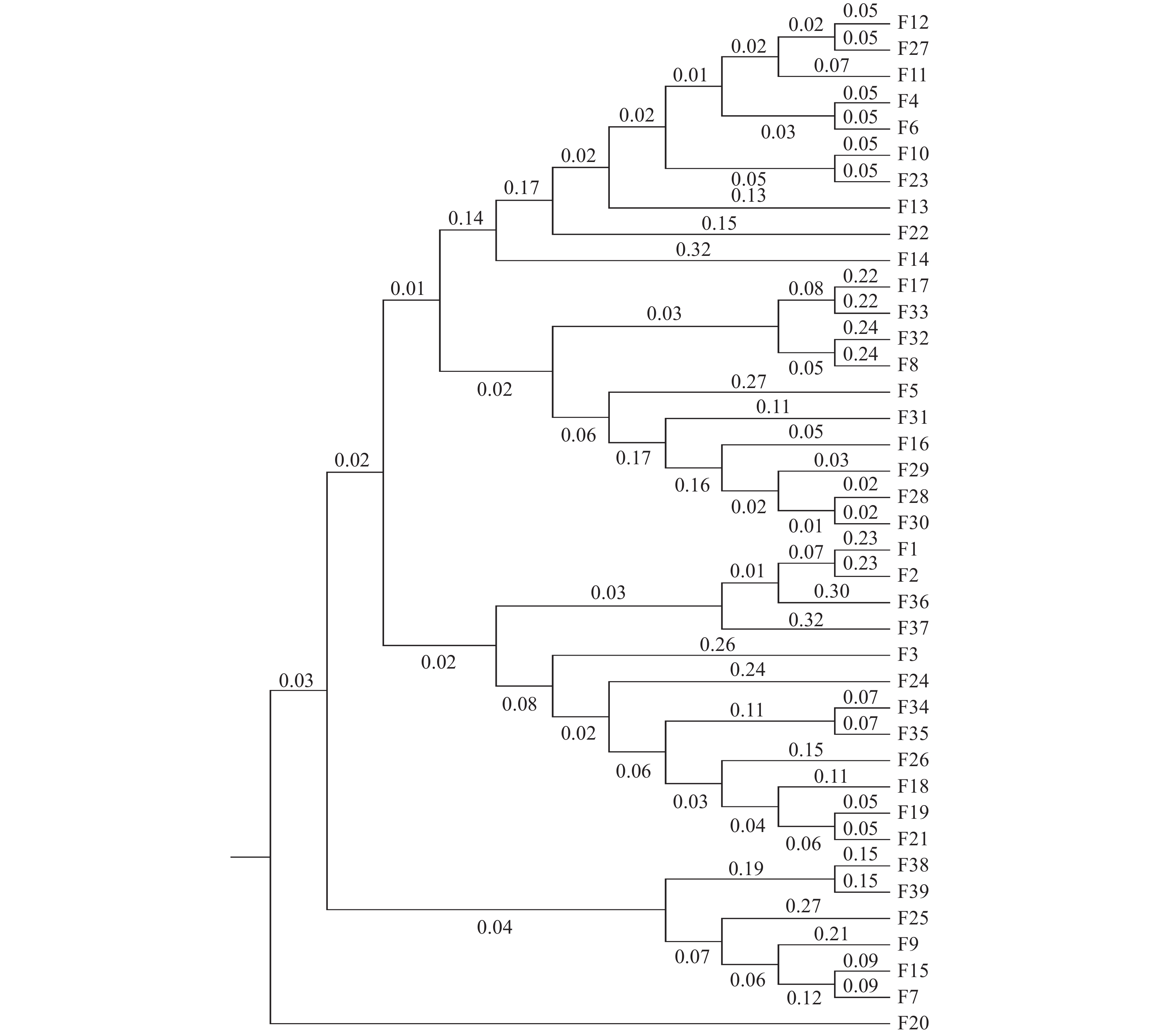
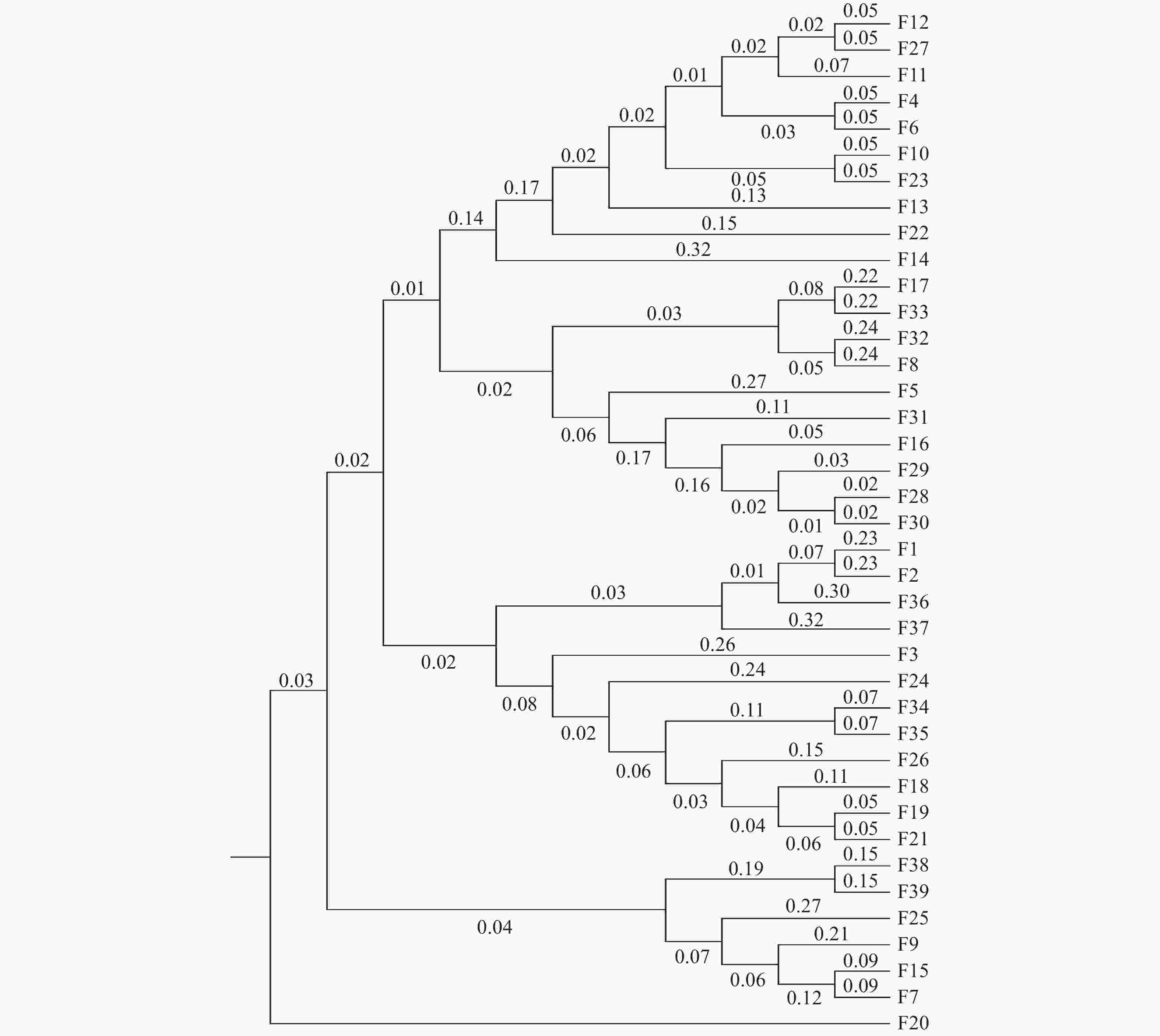
基于遗传距离的UPGMA聚类与STRUCTURE结果相似,将39个楠木家系划分4个群组(图3)。家系F20(乐山峨眉山)单独聚为一组,成都崇州、大邑、邛崃(F4、F5、F6、F8、F10、F11、F12、F13、F14、F16、F17)、乐山峨眉山(F22、F23)、泸州法王寺、纳溪(F27、F28、F29、F30、F31、F32、F33)的20个家系聚为一组,成都杜甫草堂、崇州、邛崃(F1、F2、F3、F18、F19)、乐山峨眉山(F21)、泸州法王寺(F24、F26)、眉山洪雅(F34、F35)和雅安雨城(F36、F37)的12个家系聚为一组,其余6个家系聚为一组。其中,第二组聚类、第三组聚类与STRUCTURE划分的第一类群具有相似的家系组成。
-
遗传多样性可以维持种群的可持续发展,是探究遗传结构、有效群体的重要方法 [18]。物种的遗传多样性体现了进化潜力,进行遗传多样性分析,能够为该物种遗传动态变化、适应环境能力提供指导依据,在楠木等物种的遗传资源保护方面具有重要作用[20]。
研究选取了前期筛选的14对SSR引物进行四川主要楠木种源(家系)的遗传多样性分析,根据Botstein对位点多态性程度的划分理论[21],选取的引物中MN-e96(PIC=0.306)、SSR17(PIC=0.410)为中度多态位点(0.25<PIC<0.5),其余12个多态位点均为高度多态位点(PIC>0.5),综合表现为高度多态性。结果表明选取的14对SSR引物具有多态性高、稳定性好的特点,能够顺利开展楠木分子标记研究、遗传多态性研究等相关工作。
通过Nei's遗传多样性指数(H)和Shannon信息指数(I)可以衡量各楠木种源(家系)的遗传多样性水平,供试的39个楠木家系Shannon’s 信息指数(I)平均值为1.349,观测杂合度(Ho)变化范围0.920~1.000,期望杂合度(He)变化范围0.669~0.709。说明楠木种源表现出较高的遗传多样性水平。该结果相似于Zhu[18]利用nSSR标记分析楠木育种群体得到的Shannon信息指数(I)平均值1.368,观察杂合度(Ho)0920~1.000,期望杂合度(He)0.646~0.703;不同于张炜[18]使用AFLP分子标记技术对中国主要楠木分布区采集的92个样品进行分析得到的结论:楠木遗传多样性不高,处于较低水平。这可能是因为研究材料为优良单株的半同胞家系,而张炜的研究材料为母树,这表明楠木种源内杂交较多,基因交流充分,种源后代家系的遗传多样性水平较高。分子方差分析(AMOVA)结果也显示,楠木家系的变异的主要来源是群体内(占99%),说明楠木大部分遗传差异为个体差异,基因流动频繁,降低了种群间的遗传分化[22-23]。这些结果也契合于Wang[24]研究得出的结论:林木杂交频繁导致遗传多样性较高,群体间的遗传分化较低。
楠木种源(家系)的遗传多样性体现了杂交育种是楠木遗传改良的重要方式。除了从自然群体中选择优良楠木基因型外,遗传距离在杂交试验中的亲本选择上具有指导意义,遗传距离的远近与个体的杂交优势强弱有一定联系[25]。李娜娜[26]基于遗传距离的聚类分析,应用在菊花杂交亲本选配指导中。利用聚类分析能够更清晰地揭示家系间的亲缘关系,基于遗传距离的聚类分析可将39个楠木家系划分为4个群组,家系间遗传距离在0.0721—0.9444之间;STRUCTURE遗传结构分析将楠木家系划分为3个类群。结果较清楚地呈现出了39个楠木家系的亲缘关系,总体来看聚类结果与种源地联系不明显,各类群基本均混合了各种源地的家系,这可能是由于选择的种源均位于四川省,母树以庙宇、乡村四旁保存的古树为主,为人工栽植,亲缘关系较为复杂,该结果说明在后续楠木杂交育种研究中,需根据遗传背景信息进行亲本选择,不能单纯以种源地进行亲本选择,呈现的四川楠木种源(家系)的聚类分析结果为楠木后续杂交育种亲本选择奠定了基础。
-
利用前期开发的14对SSR分子标记对四川省10个主要楠木种源39株母树的半同胞家系进行遗传多样性和遗传结构分析。通过14对SSR引物共检测出178个等位基因(Na),平均有效等位基因数(Ne)为7.058,PIC值在0.306~0.939间变化,均值为0.746,多态性较高。楠木种源(家系)的Shannon’s信息指数(I)在1.306~1.511之间变化,平均值为1.368,表明楠木种源(家系)具有较高的遗传多样性水平。期望杂合度(He)与观测杂合度(Ho)分别在0.646~0.0.703之间和0.920~1.000之间,平均分别为0.676和0.550。分子方差分析(AMOVA)显示99%的遗传变异来源于种源内,仅1%的遗传变异来源于种源间,表明楠木种源内杂交较多,种源后代家系的遗传多样性水平较高。基于STRUCTURE对39个家系进行群体结构分析,当K=3时,ΔK有最大峰值,表明适宜被分为3个群组。基于遗传距离的聚类分析将楠木39个家系分为4个类群。类群划分与种源地无明显联系,说明后续楠木杂交育种研究中,需根据遗传背景信息进行亲本选择,不能单纯以种源地进行亲本选择。研究结果较清楚地呈现出了39个楠木家系的亲缘关系,为楠木优异基因资源的发掘和杂交亲本的选配提供了理论参考。
Genetic diversity of Phoebe zhennan Provenances (Families) from Sichuan Province based on SSR markers
doi: 10.12172/202401030001
-
Key words:
- Pheobe zhennan /
- family /
- SSR molecular marker /
- genetic diversity
Abstract: Pheobe zhennan is a unique and valuable timber tree plants in China. In order to reveal the genetic background and relationship among families, and to provide theoretical basis for the utilization of P. zhennan, 14 pairs of polymorphic SSR primers were used to analyze the genetic diversity of 39 P. zhennan families from 10 provenances in Sichuan province. The results showed that a total of 178 alleles (Na) were detected by 14 SSR primers, and the average effective number of alleles (Ne) was 7.058. Shannon's information index (I) ranged from 1.306 to 1.511, with an average value of 1.368, reflected a relative high level of genetic diversity of the P. zhennan provenances (families). The expected heterozygosity (He) and observed heterozygosity (Ho) ranged from 0.646 to 0.703 and from 0.920 to 1.000, with an average of 0.676 and 0.550, respectively. The PIC values ranged from 0.306 to 0.939, with an average value of 0.746, showing high polymorphism of the SSR markers. The result of molecular analysis of variance (AMOVA) showed that 99% of genetic variation came from within provenances, only 1% came from inter-provenances. The genetic structure was determined by STRUCTURE software, when K=3, the ΔK had the maximum peak value, indicating that it was suitable to be divided into three groups. The cluster analysis based on genetic distance also divided 39 P. zhennan families into three groups. Except for F20, the other three groups were mixed with other nanmu families from Chengdu, Luzhou, Ya 'an and Leshan, and there was no obvious relationship between the division of groups and provenance sites. The results of this study provided a theoretical reference for the exploration of excellent gene resources and the selection of hybrid parents.




















 DownLoad:
DownLoad: