-
成都麻羊,又名四川铜羊,原产于成都平原及附近丘陵地区,是我国优良肉用地方山羊品种[1, 2]。成都麻羊具有生长发育快、体格较大、性早熟、繁殖能力强、屠宰率高、膻味轻、肉质嫩、板皮优良、肉皮兼用等显著特点,现有丘陵型和山地型两个生态类型[1-3]。在我国,早有引进成都麻羊改良当地山羊培育新品种的案例,如四川省有名的南江黄羊、金堂黑山羊品种[1-3]。但是,圈养规模较小、缺乏科学的管理、近亲繁殖现象等问题会导致成都麻羊的遗传变异丢失[4],引起圈养种群退化,长期会导致遗传多样性逐渐丧失,一些特有的重要基因资源会在培育中消失殆尽,同时会造成个体适应能力变弱,抗病力差,死亡率增加等[5],长期下来会影响圈养成都麻羊种群的健康发展。畜禽遗传资源本质上是基因资源,保护畜禽遗传资源就是保护基因的多样性[6]。因此,成都麻羊作为我国山羊品种的一个宝贵基因库,维持其种群遗传多样性,最大限度地降低遗传损失,保证其种群遗传健康,对其种群种质资源的良性发展和种群的遗传管理都具有重要意义。
微卫星DNA(microsatellite DNA),是以少数几个核苷酸为基本单位进行多次串联重复的DNA序列,因而也被称为简单序列重复(simple sequence repeat,SSR)或短串联重复序列(short tandem repeat,STR)[6-9]。相比其他的分子标记技术,微卫星DNA标记优点突出,如呈现共显性遗传、高度多态性等,是目前最为准确、先进的遗传标记系统之一,是种群遗传结构研究和多样性评估最常用的工具[5, 7, 10-14]。在山羊、绵羊中,利用微卫星DNA标记测定遗传多样性已有较多相关研究[9, 11, 15-21]。
鉴于此,为了加强成都麻羊资源的保护和利用,实现优质地方山羊品种种质资源的良性开发,满足市场对成都麻羊的发展需求,借助微卫星DNA作为遗传标记,对成都麻羊圈养种群进行遗传多样性现状调查,以期为该资源群体的遗传背景研究提供分子理论依据,同时对合理利用成都麻羊资源和可持续发展也具有重要意义。
HTML
-
本研究以成都西岭雪农业开发有限公司(四川省成都市大邑县)饲养的成都麻羊为研究对象,共随机挑选268只个体采集血液样品,其中雄性个体18只,雌性个体250只。对每只成都麻羊个体进行颈静脉采血5 mL,随后用EDTA抗凝混匀,置于冰盒中带回实验室,−20 ℃保存。
-
本研究采用Tianamp基因组DNA提取试剂盒提取DNA,提取步骤参照使用说明书执行。将提取得到的DNA用1%的琼脂糖凝胶进行电泳检测,并将其保存于−20 ℃冰箱。
参考已发表文献[7, 9, 10, 16, 17, 20, 21],共选46对多态性微卫星引物委托生工生物工程(上海)股份有限公司合成。通过预实验筛选,有9对引物(见表1)能较稳定地扩增得到多态性较高的微卫星位点。PCR扩增体系(20 uL):DNA模板(浓度25 ng/uL)2 uL,正向引物(浓度10 umol/ul)0.5 uL,反向引物(浓度10 umol/ul)0.5 uL,Mix溶液10 uL,灭菌双蒸水7 uL。PCR扩增条件:首先95 ℃预变性持续15 min;之后94 ℃变性持续30 s,相应退火温度退火持续30 s,72 ℃延伸持续30 s,并设置30个循环;最后72 ℃终止延伸持续10 min。每次扩增过程中均设定阴性对照,每个样品扩增3次。PCR扩增产物经1.5%的琼脂糖凝胶电泳检测后拍照。
微卫星位点 引物序列(5′-3′) 5′端修饰 退火温度/℃ ILSTS004 F: CTTAAAATCTGTCTTTCTTCC 5`6-FAM 48 R:TAGTGTGTATTGGTTTCTCC INRA063 F: ATTTGCACAAGCTAAATCTAACC 5`HEX 52 R:AAACCACAGAAATGCTTGGAAG OarFCB011 F: GCAAGCAGGTTCTTTACCACTAGCACC 5`6-FAM 54 R:GGCCTGAACTCACAAGTTGATATATCTATCAC ILSTS030 F: CTGCAGTTCTGCATATGTGG 5`HEX 54 R:CTTAGACAACAGGGGTTTGG SR-CRSP-1 F: TGCAAGAAGTTTTTCCAGAGC 5`HEX 54 R:GTACCCTGGTTTCACAAAAGG SR-CRSP-5 F: GGACTCTACCAACTGAGCTACAAG 5`6-FAM 54 R:TTATGAAATGAAGCTAAAGCAATGC ILSTS033 F: TATTAGAGGATGGATGCTAAACT 5`HEX 52 R:CTGCAAATAAGAAAACTGAATAAA OarFCB48 F: GAGTTAGTACAAGGATGACAAGAGGCAC 5`HEX 54 R:GACTCTAGAGGTACGCAAAGAACCAG McM038 F: TGGTGAATGGTGCTCTCATACCAG 5`HEX 54 R:CAGCCAGCAGCCTCTAAAGGAC Table 1. Microsatellite DNA markers were used in this study
-
使用GeneMarker V2.2.0(Demo)软件,根据DNA片段大小范围和峰形质量确认等位基因。3个重复试验分型一致的结果直接录入数据库,若结果有差异,则需要重新跑PCR甚至重新提取DNA。利用 Excel 2007软件整理所有样品对应微卫星位点的等位基因型数据。
-
利用Micro-Checker[22]检验每个位点是否存在无效等位基因或等位基因缺失等情况。利用Genepop on the Web[23]和GenAlEx[24]软件,对每个微卫星位点是否偏离哈迪-温伯格平衡(Hardy-Weinberg equilibrium,HWE)和是否存在连锁不平衡(Linkage disequilibrium,LD)进行分析。哈迪-温伯格平衡检测中,若P>0.05,则符合哈代-温伯格平衡,说明这个群体是随机交配群体;若P<0.05,则呈显著偏离哈代-温伯格平衡状态;若P<0.01,则呈极显著偏离哈代-温伯格平衡状态。利用Cervus、GenAlEx、Excel Microsatellite Toolkit统计各个微卫星标记位点的等位基因数(k)、有效等位基因数(Ne)、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)和多态信息含量(Polymorphism information content,PIC)[24, 25]。当PIC<0.25、0.25<PIC<0.5和PIC>0.5时,分别表示该座位为低度、中度和高度多态位点,其中高度多态位点能够提供大量的遗传信息,中度多态位点可以提供较为合理的信息[10, 11, 13, 20, 21, 26]。
1.1. 研究材料
1.2. 研究方法
1.2.1. 基因组DNA提取与检测
1.2.2. 基因分型
1.2.3. 数据分析
-
本研究筛选得到的9对多态性微卫星位点中,高度多态标记位点有5对(PIC>0.5),可提供大量的遗传信息,中度多态标记位点有3对(0.25<PIC<0.5),能够提供较为合理的信息,低度多态标记位点仅1对(PIC<0.25),为SR-CRSP-1,可提供的信息量较差(见表2)。9个微卫星标记位点的等位基因数范围是3~7,平均值是4.89;其中,等位基因数是4及以上的微卫星位点有7个,占多态性微卫星位点总数的77.8%;等位基因数在5~7之间的微卫星位点有6个,占多态性微卫星位点总数的66.7%;仅有2个微卫星位点的等位基因数是3个(见表2)。观察杂合度的变异范围是0.250~0.839,平均值是0.547;期望杂合度的变异范围是0.226~0.766,平均值是0.539(见表2)。多态信息含量的变异范围是0.226~0.766,平均值是0.515(见表2)。
依据哈代-温伯格平衡检测中的P值,9个微卫星标记位点中有7个标记位点符合哈代-温伯格平衡,占77.8%;另外2个标记位点(INRA063和SR-CRSP-5)显著偏离哈代-温伯格平衡,占22.2%。
依据连锁不平衡检测结果,9个微卫星标记位点之间均不存在连锁不平衡现象(P>0.001),表明所选用的微卫星标记位点是相互独立的遗传标记系统。
-
排除SR-CRSP-1外之后,使用剩余的8个微卫星标记位点对圈养成都麻羊种群进行遗传多样性分析,共有60个等位基因被成功检测,各标记位点的等位基因数介于5~11之间,平均值为7.5;各标记位点的平均有效等位基因数为2.36个;各个位点私有等位基因检测并未发现其存在;观察杂合度的变异范围是0.413~0.770,平均值是0.577;期望杂合度的变异范围是0.493~0.777,平均值是0.634;多态信息含量的变异范围是0.432~0.743,平均值是0.580(见表3)。
微卫星位点 k N Ho He PIC F(Null) ILSTS004 5 31 0.452 0.435 0.404 −0.0615 INRA063 5 31 0.516 0.654 0.598 0.1032 OarFCB011 4 32 0.594 0.629 0.551 −0.0297 ILSTS030 6 32 0.813 0.685 0.633 −0.0991 SR-CRSP-1 3 32 0.250 0.226 0.226 −0.0608 SR-CRSP-5 7 32 0.594 0.591 0.591 −0.0116 ILSTS033 5 29 0.414 0.459 0.459 0.0122 OarFCB48 6 31 0.839 0.766 0.766 −0.0582 McM038 3 31 0.452 0.410 0.410 −0.0721 注:k表示等位基因数,N表示DNA样品量,Ho表示观察杂合度,He表示期望杂合度,PIC表示多态信息含量 Table 2. Polymorphism detection results of 9 microsatellite markers
微卫星标记 k N Ho He PIC HWE F(Null) ILSTS004 11 260 0.565 0.610 0.585 NS 0.0367 INRA063 5 254 0.594 0.675 0.622 NS 0.0579 OarFCB011 8 260 0.531 0.635 0.565 NS 0.0900 ILSTS030 7 267 0.704 0.696 0.656 NS −0.0110 SR-CRSP-5 8 261 0.598 0.614 0.567 NS 0.0063 ILSTS033 7 262 0.443 0.491 0.468 NS 0.0500 OarFCB48 9 256 0.770 0.777 0.743 NS 0.0037 McM038 5 264 0.413 0.493 0.432 NS 0.0815 注:k表示等位基因数,N表示DNA样品量,Ho表示观察杂合度,He表示期望杂合度,PIC表示多态信息含量,HWE表示哈代-温伯格平衡 Table 3. Genetic diversity of captive Chengdu gray goat population
-
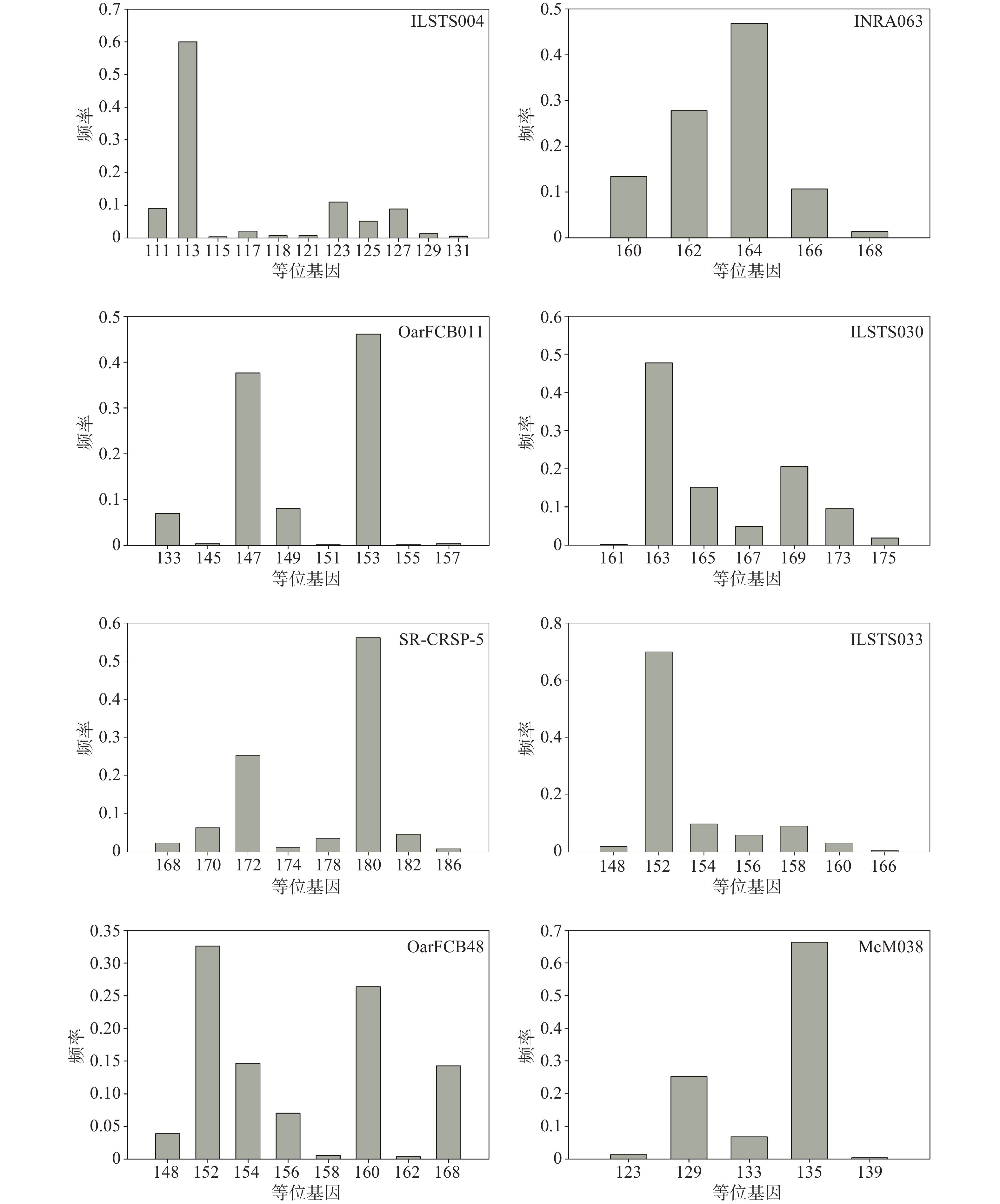
在8个微卫星标记位点中,等位基因较多的前4个是:ILSTS004,共检测到11个等位基因,大小分布在111~131 bp,频率范围为0.0038~0.6000;OarFCB48,共检测到9个等位基因,大小分布在148~168 bp,频率范围为0.0039~0.3262;SR-CRSP-5,共检测到8个等位基因,大小分布在168~186 bp,频率范围为0.0077~0.5613;OarFCB011,共检测到8个等位基因,大小分布在133~157 bp,频率范围为0.0019~0.4615(见图1)。
在8个微卫星标记位点中,等位基因较少的4个位点是:ILSTS033,共检测到7个等位基因,大小分布在148~166 bp,频率范围为0.0057~0.6985;ILSTS030,共检测到7个等位基因,大小分布在161~175 bp,频率范围为0.0019~0.4775;McM038,共检测到5个等位基因,大小分布在123~139 bp,频率范围为0.0038~0.6629;INRA063,共检测到5个等位基因,大小分布在160~168 bp,频率范围为0.0138~0.4685(见图1)。
2.1. 微卫星位点检测
2.2. 种群遗传多样性分析
2.3. 微卫星标记位点等位基因分布
-
遗传多样性是一个物种适应环境变化所必需的条件[7]。种群内的遗传多样性反映了一个物种的进化潜力,是其遗传特征进化的基础[26]。一个物种的遗传变异越丰富,其对自然选择和环境变化的适应能力就越强[6, 9, 12, 27]。遗传多样性通常用等位基因多样性、多态性、平均杂合度等指标来描述[6, 12, 16, 19]。
在同一微卫星位点中,随着核心重复序列重复次数的变化,该位点的片段长度也会发生变化,继而形成等位基因[11, 17];等位基因是形成群体遗传多态性的基础,等位基因数越多,遗传多态性越丰富[17]。评价位点多态性高低的指标是多态信息含量[17]。本研究从9个微卫星位点中筛选到5个高度多态的位点、3个中度多态性的位点、1个低度多态性的位点,因此仅有前8个可作为有效的分子标记进行群体遗传多样性分析。在后续的分析中我们使用了这8个中高度多态的微卫星位点分析了268份成都麻羊样品,最终在这8个位点中一共检测到的等位基因数为60个,平均等位基因数为7.5,与其他绵羊种群相比,要高于甘肃高山细尾羊、滩羊、藏羊等[7],低于山东省的小尾寒羊、大尾寒羊、山地绵羊、洼地绵羊等[28],与四川省布拖黑绵羊种群基本持平[10],但与其他山羊种群相比,要高于山西黎城大青羊、山西吕梁黑山羊、宁夏中卫山羊、贵州白山羊、云南威信白山羊等[11, 16, 19],低于湖南湘东黑山羊、重庆川东白山羊、云南云岭山羊、河南黄淮山羊等[11]。不过,这并不能排除种群大小和所用微卫星位点数不同而造成的差异。有研究表明,如果样本量太少,会导致一些频率较低的等位基因无法被检测到,进而对试验结果的准确性造成影响[9]。本研究使用了8个微卫星位点分析了268份成都麻羊样品,微卫星位点数量属于平均水平,种群样本量较大,而个别其他研究的样本量最少仅有20余份[11, 17],微卫星位点数量最少仅有5个[7, 17]。以同一微卫星位点而言,种群样本量越大,其观察到的等位基因数往往越多,以微卫星位点ILSTS004、OarFCB011和ILSTS030为例,本研究中观察到的等位基因数目分别为11、8和7,而在刘成建的研究中(涉及20个样本量)仅为5和4[17]。此外,在本项研究中,成都麻羊群体中8个微卫星位点的多态信息含量的变异范围是0.432~0.743,平均多态信息含量0.580,与刘成建的研究结果(0.604)基本一致[17],表明了成都麻羊群体具有较丰富的遗传多样性。
基因杂合度,它反映了群体在几个位点的遗传变异情况,是被检测的遗传标记的多态性的度量参数[10, 17]。若某个群体的基因杂合度大于0.5,则意味着该群体还未受到高强度的选择压力,其遗传多样性比较丰富[10, 17]。在本项研究中,成都麻羊群体的平均观察杂合度和平均期望杂合度分别为0.577和0.634,表明该群体有较丰富的遗传多样性,该结果与前述多态性的分析结果相一致。另外,刘若余等人基于线粒体D-loop序列调查了中国9个山羊品种的遗传多样性,其中成都麻羊的单倍型多样性和核苷酸多样性分别为1.0000±0.0243和0.017956±0.004410,同样表明其有着丰富的遗传多样性[29]。
有效等位基因数也可以反应群体的遗传变异大小,在数值上等于基因纯合度的倒数[9, 16, 17]。若有效等位基因数越接近所检测到的等位基因的绝对数,则说明等位基因在群体中分布越均匀[9, 16, 17, 21]。本研究中,成都麻羊群体平均有效等位基因数(2.36)低于实际观测到的等位基因数(7.5),说明有些等位基因频率相对较高,而另一些相对较低,等位基因在群体中分布得并不均匀,图1也具体地展示了这一现象,究其原因应该是圈养种群频繁人工选择和一定的近亲繁殖造成的。研究同时发现,成都麻羊群体的观察杂合度和期望杂合度之间差异很小,仅为0.057,这说明就目前而言,成都麻羊群体受外来选择压力和近交繁殖等因素的影响还比较小,群体依旧处于遗传平衡的状态
种群遗传学管理的目的是保持种群的遗传多样性,避免近亲繁殖[30]。如果对一个封闭的小种群不采取任何人工干预措施,最终必将造成近亲交配,进而引发近交衰退。鉴于成都麻羊种群的遗传多样性还比较高,受近亲繁殖等因素影响还较小,具有较高的保种潜力,建议开展全面采样,借助微卫星DNA标记技术,完善个体信息档案,构建起具有完整代表性的成都麻羊种群遗传谱系关系。



















 DownLoad:
DownLoad: